实际测序任务中,您可能期望使用阿里云AGS Remote Api来实现Mapping流程,而获取Bam数据后续的Call操作期望在自己集群实现,本方案将讨论如何使远程的API调用和专有集群中的后续流程结合使用。
系统原理:
整个测序流程通过一个本地AGS工作流完成:工作流中嵌入一个或多个task,并调用Remote Api完成Fastq的Mapping流程;获取Bam文件后,本地工作流来完成后续测序任务。
流程解读:
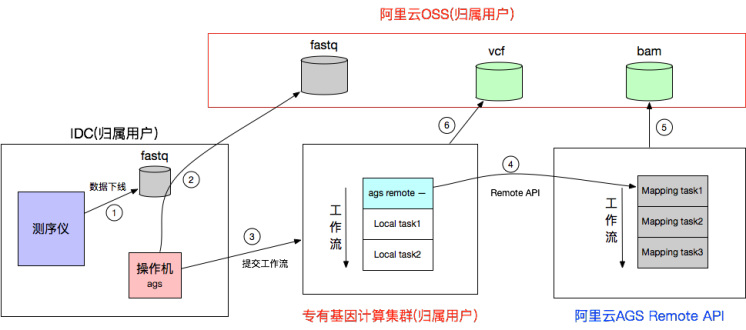
上述系统包括几个部分:
用户IDC环境:包括测序仪系统、本地数据缓存、操作机;
用户的阿里云基因集群:用户在ACK中申请的K8S专有集群;
阿里云RemoteApi系统:通过API提供Mapping服务;
阿里云OSS服务:用于保存用户源数据、中间Bam数据、最终的vcf数据;
操作流程:
- 用户IDC环境内,基因数据下线暂存;
- 用户通过数据传输工具(Ossutil)将待处理的fastq数据上传到阿里云OSS存储服务(用户账号下的OSS);
- 用户通过AGS命令行工具,像用户的专有集群提交测序服务;
- 测序工作流的task1:向阿里云AGS服务提交RemoteApi请求,执行fastq数据的Mapping服务,task1等待mapping服务完成后再执行后面的任务;
- AGS Mapping服务会下载fastq数据,对比完成后会将bam数据上传到oss;
- 用户工作流完成剩余的任务,下载bam文件、执行call变异、上传最终数据等;
环境准备:
- AGS命令行下载地址:
详见:https://help.aliyun.com/document_detail/148762.html
- OSS数据授权:
详见:https://help.aliyun.com/document_detail/148762.html
- Fastq数据上传
详见:https://help.aliyun.com/document_detail/148762.html
任务提交:
提交混合方式的工作流,需要您编辑自己的工作流YAML模板,示例如下:
1. 模板示例:
具体应用需要根据业务进行定制,例如:
使用什么RemoteApi接口?
在什么位置调用?
具体的调用参数是什么?
在专有集群中执行什么任务?
等等,
下面示例,描述如何在一个工作流中即包含RemoteApi的调用,并进行专有集群任务的调用:
apiVersion: argoproj.io/v1alpha1
kind: Workflow #new type of k8s spec
metadata:
generateName: mpileup- #name of workflow spec
spec:
entrypoint: mpileup #invoke the whalesay template
arguments:
parameters:
- name: AKID
value: ""
- name: AKSEC
value: ""
- name: OSSURL
value: "oss-cn-shenzhen-internal.aliyuncs.com"
# fastq define
- name: region
value: "cn-shenzhen"
- name: bucket
value: "shenzhen"
- name: fastq1
value: "fastq/MGISEQ2000/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_1.fq.gz"
- name: fastq2
value: "fastq/MGISEQ2000/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_2.fq.gz"
# gene define
- name: bamFile
value: "output/bam/gene.bam"
- name: cpuNumber
value: "32"
- name: vcfFileName
value: "gene.vcf"
- name: service
value: "s"
templates:
- name: agsmapping
container:
image: registry.cn-hangzhou.aliyuncs.com/aliyun-gene-service/ags-cli:v1.0.1-a5a06af
imagePullPolicy: Always
command: [bash,-c]
args:
- region={{workflow.parameters.region}};
akID={{workflow.parameters.AKID}};
akSec={{workflow.parameters.AKSEC}};
akSecAuth=`echo -n $akSec | base64`;
mkdir -p /root/.ags; echo "{"region":"cn-beijing","access_key_id":"$akID","access_key_secret":"$akSecAuth","cluster_id":"","kubectl_location":""}" > /root/.ags/config;
ags remote run mapping --region $region --fastq1 {{workflow.parameters.fastq1}} --fastq2 {{workflow.parameters.fastq2}} --bucket {{workflow.parameters.bucket}} --output-bam {{workflow.parameters.bamFile}} --markdup "true" --service {{workflow.parameters.service}} --reference=hg19 &> ags.log;
sucessline=`cat ags.log | awk -FJobName '{print $2}' | awk -F" '{print $3}'`;
if [ "$sucessline" = "" ]; then
echo "ags submit with error "$res;
exit 1;
fi;
taskName=${sucessline:0:-1};
for ((i=0; i<100; i++));
do
success=`ags remote get $taskName | grep Succeeded | wc -l`;
if [ "$success" = "1" ]; then
echo "ags task successful";
break;
fi;
echo "exec command ags remote get $taskName";
sleep 60;
done;
- name: mpileupprepare
container:
image: registry.cn-shenzhen.aliyuncs.com/plugins/gene-tools:ags-hunhe
imagePullPolicy: Always
command: [sh,-c]
args:
- ossutil config -e {{workflow.parameters.OSSURL}} -i {{workflow.parameters.AKID}} -k {{workflow.parameters.AKSEC}};
bamFile={{workflow.parameters.bamFile}};
bamRootDir=/data;
mkdir -p $bamRootDir/$bamFile;
ossutil cp --parallel=100 oss://{{workflow.parameters.bucket}}/$bamFile $bamRootDir/$bamFile;
samtools view $bamRootDir/$bamFile/`basename $bamFile`;
resources:
requests:
memory: 2Gi
cpu: 2
- name: mpileup
dag:
tasks:
- name: agsmappingtask
template: agsmapping
- name: tasks-prepare
dependencies: [agsmappingtask]
template: mpileupprepare过ags命令行工具提交工作流,参考文档:https://help.aliyun.com/document_detail/121342.html
工作流编排模板,一般可以有两部分:RemoteApi部分 + 专有集群任务部分;
例如编排模板可以有如下任务:
任务1:调用RemoteApi执行fastq数据的mapping流程,并等待mapping结束;
任务2:下载任务1生成的数据,并执行samtools view处理命令;
其中:registry.cn-hangzhou.aliyuncs.com/aliyun-gene-service/ags-cli:v1.0.1-a5a06af 为包含了ags命令行工具的镜像,可以直接使用。
2. Remote Api命令解析:
AGS RemoteAPI提交命令如下:
ags remote run mapping
--region cn-shenzhen # region of oss, e.g. cn-shenzhen, cn-beijing and etc
--fastq1 MGISEQ/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_1.fq.gz # filename of fastq pair 2, fastq-pathfilename
--fastq2 MGISEQ/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_2.fq.gz # filename of fastq pair 1
--bucket my-test-shenzhen # Bucket name
--output-bam bam/MGISEQ_NA12878_hs37d5.bam # Output filename of BAM
--service "g" #SLA: [n:normal|s:silver|g:gold|p:platinum]
--markdup [true|false|default true] #Mark Duplicated, by default true
--reference [hg19|hg38|<reference path on OSS>]service:提供服务等级,n为普通模式,处理速度较慢;s、g、p提供的服务使用更小的时间;
